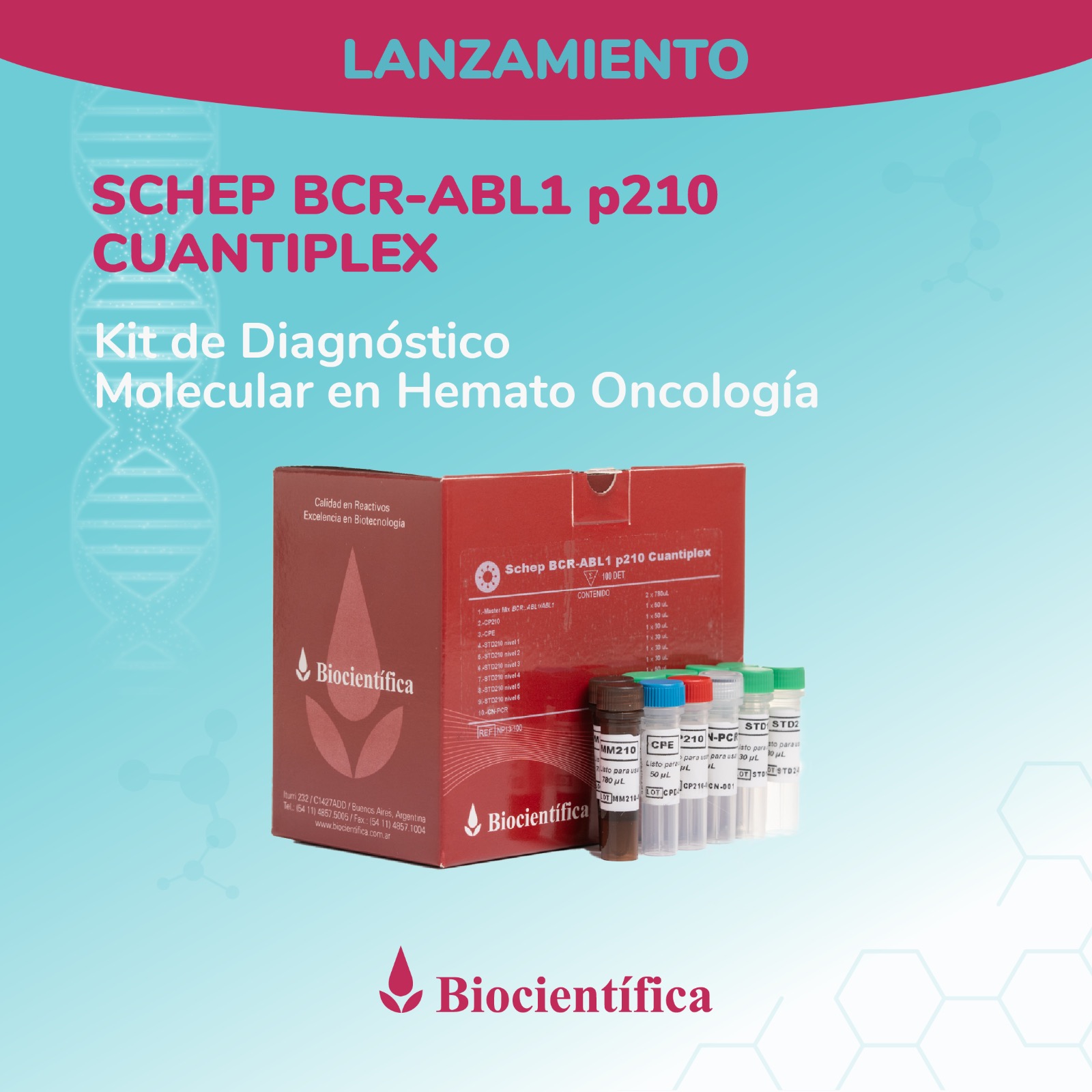
Argentino de Diagnóstico
Molecular en Hemato Oncología!
para mejorar la calidad de vida
para mejorar la calidad de vida
Schep Dengue Screen
en tecnología de PCR en Tiempo Real,
aprobado por ANMAT.
para la detección rápida de SARS-CoV-2
¡Ahora en formato Multiplex y en un solo paso!
Desarrollamos, producimos, comercializamos en Argentina y exportamos reactivos de diagnóstico in vitro (IVD) y productos de investigación (RUO). Además, importamos y comercializamos en Argentina reactivos y equipamiento de laboratorio, investigación biológica, agrobiotecnología (salud animal, plantas y semillas), estudios forenses, estudios de suelos y alimentos.
Desarrollamos la línea Schep para diagnóstico por PCR en Tiempo Real, actualmente con aplicaciones para diagnóstico del virus del dengue, SARS-CoV-2, Influenza A y B y monitoreo de leucemia.
Categorías de Productos

DIAGNÓSTICO IN VITRO
Productos de diagnóstico in vitro para enfermedades humanas. Nuestro portfolio incluye desarrollos propios y las mejores marcas internacionales
Conocé nuestro catálogo para Argentina y para exportación.

INVESTIGACIÓN BIOLÓGICA
Productos para investigación biomédica, estudios forenses, biotecnología para el agro, veterinaria y alimentos.
Las mejores marcas de Life Sciences disponibles sólo para Argentina.
Garantía de calidad
Nos comprometemos con la mejora continua de nuestros productos y procesos. Conocé nuestro Sistema de Garantía de Calidad.
Servicio y postventa
Priorizamos la satisfacción de nuestros clientes con un portfolio de productos de excelencia, soporte técnico y atención personalizada.
Comunidad y RSE
Construimos comunidad. Brindamos servicios de capacitación y actualización para bioquímicos e investigadores en biociencias. ¡Sumate!
Redes de distribución
Contamos con una red de comercialización en toda la Argentina. Exportamos a 20 países en el mundo.
Nuestros clientes
– Laboratorios de análisis clínicos.
– Laboratorios de anatomía patológica.
– Laboratorios de análisis de alimentos.
– Centros de epidemiología y enfermedades infecciosas.
– Institutos de I+D y universidades.
– Industria farmacéutica y biotecnológica.
– Industria petroquímica.
– Empresas semilleras.
– Hospitales, sanatorios y clínicas.
¿Te gustaría recibir información de la industria y novedades de biocientífica?
Suscribite ahora a nuestro Newsletter
Marcas Representadas